Variants du SARS-CoV-2
Le SARS-CoV-2 (coronavirus 2 du syndrome respiratoire aigu sévère) est le virus qui cause la maladie à coronavirus 2019 (Covid-19). La séquence WIV04 / 2019 serait la séquence d'origine infectant les humains. Comme tout virus, le SARS-CoV-2 a évolué et a créé des variants. Certains sont considérés comme d'une importance particulière. D614G est probablement la mutation la plus importante et l'une des plus répandues du SARS-CoV-2. Cette mutation empêche un mécanisme d'infection des globules blancs connu sous le nom de facilitation de l'infection par des anticorps.
Les coronavirus sont censés être stables dans la mesure où ils produisent une enzyme correctrice d’erreurs appelée « exoribonucléase ». D’après l’INSERM, le SARS-CoV-2 muterait ainsi deux fois moins rapidement que les virus grippaux. Dans les faits, le SARS-CoV-2 se révèle assez mutagène.

Fin 2020 et début janvier 2021, trois variants problématiques sont identifiés : le variant alpha en Angleterre, le variant bêta en Afrique du Sud, et le variant gamma au Brésil. En 2021, au moins sept autres variants ont été recensés par l’OMS comme d’intérêt. Les variants les plus contagieux (de 50 à 75 % par rapport à la souche initiale) ont été successivement le variant alpha durant l', puis le variant delta identifié en Inde à partir du .
Fin 2022, de nouveaux variants dérivant de la souche Omicron circulent dans le monde. : BQ.1.1 en Europe ou XBB en Asie sont "surveillés du fait de leurs mutations sur la protéine Spike, leur conférant un potentiel pouvoir d’échappement immunitaire".
Ces variants ne remettent pas en cause l’efficacité des vaccins contre la Covid-19. Seules des combinaisons de mutations bien spécifiques sur la protéine S sont susceptibles en théorie de compromettre l'efficacité des vaccins. Une combinaison déjà identifiée pourrait être le trio de mutations : F140-, Y248* et E484K.
Nomenclature
Aucune nomenclature pour les lignées évolutives du SARS-CoV-2 n'est universellement acceptée, cependant en , l’Organisation mondiale de la santé travaille sur une « nomenclature standard pour les variants du SARS-CoV-2 qui ne fait pas référence à une localisation géographique ». Dans une lettre d'information datée du , l'OMS annonce nommer les variants par une lettre grecque. L'OMS précise que « ces noms ne remplacent pas les noms scientifiques [attribués par d'autres], qui véhiculent des informations scientifiques importantes ».
Bien qu'il existe plusieurs milliers de variants du SARS-CoV-2, les sous-types du virus peuvent être placés dans des groupes beaucoup plus larges tels que les lignées ou les clades. Plusieurs nomenclatures différentes pour ces sous-types ont été proposées.
Catégorisation des variants
- variant préoccupant, VOC (variant of concern)
- variant d'intérêt, ou variant à suivre, VOI (variant of interest) également dénommé VUI (variant under investigation)
- variant en cours d’évaluation, ou variant sous surveillance, VUM (variant under monitoring)
Différentes nomenclatures
Entités réalisant un nommage
Nextstrain
Nextstrain nomme les clades majeurs par le numéro de l'année et une lettre (les lettres sont successives, depuis « A », chaque nouvelle année), par exemple : 19A, 19B, 20A. Pour un clade émergent, la notation comporte alors le nom du clade parent, et la mutation de nucléotide qui le définit (attention, la numérotation est par nucléotide, et non par codon), par exemple, 19A/28688C signifie : pour le clade 19A, en position de nucléotide 28688, la base nucléique est maintenant la cytosine (C). Une fois que le clade émergent a atteint les critères nécessaires, il est renommé comme clade majeur, par exemple : 19A/28688C devient 20D.
Les règles de nommage ont été révisées début pour éviter les nommages géographiques tel 20A.EU1 (variant émergent en Europe). Une notation mixte 20H/501Y.V2 ou 20I/501Y.V1 est alors utilisée avec un numéro d'ordre : V1, V2, etc.
Lors de cette révision des règles, les noms des clades ont été conservés (rétrocompatibilité), sauf pour trois noms :
- 20A.EU1 devient « 20E (EU1) » – 20A.EU1 ayant été élevé au rang de clade (il devrait alors se nommer « 20E » uniquement) ; les parenthèses « (EU1) » établissent le lien entre l'ancien et le nouveau nom ;
- 20B/501Y.V1 devient 20I/501Y.V1 – nommage plus complexe : 20B/501Y.V1 ayant été élevé au rang de clade (nommé « 20I » uniquement), mais ayant été précédemment défini avec le nom « 501Y.V1 » seul, cette même terminaison a été conservée, sans provoquer de confusion ;
- 20C/501Y.V2 devient 20H/501Y.V2 – idem.
Le nom d'un clade émergeant s'effectue avec le nom du clade parent et, selon le numéro de nucléotide, la mutation qui le définit, par exemple : 20G/1927C signifie un changement de nucléotide (ici en cytosine – C) à la position 1927, dans le clade 20G. Cependant, afin de pouvoir nommer des mutations au niveau des codons, une règle a été définie pour attribuer des noms par mutation dans chaque cadre de lecture ouvert (ORF) codant chaque protéine, par exemple 20B/S.484K signifie une mutation du codon (ici codage de la lysine – K) à la position 484 dans la protéine S (notation « S. ») du virus. Les numéros de codons sont relatifs à chaque ORF.
Depuis les noms attribués par l'OMS sont alors indiqués :
Depuis , les lignées Pango sont également indiquées :
GISAID
En mars 2021, GISAID avait défini huit clades pour les variants du SARS-CoV-2. Les clades sont nommés d'après leur mutation essentielle. Depuis les deux clades initiaux, « S » et « L » (ces noms proviennent d'une étude préalable à celle de GISAID), les autres clades sont :
- S
- L
- V (pour NS3-G251V)
- G (pour S-D614G)
- GR (pour N-G204R)
- GRY (pour S-N501Y)
- GH (pour NS3-Q57H)
- GV (pour S-A222V)
- GR (pour N-G204R)
Ces clades GISAID peuvent alors être affinés par la définition de lignées, comme celles de Lignées Pango.
Lignées Pango
Le système de nomenclature « Lignées Pango » est un système standardisé et dynamique, désignant les variants alarmants du SARS-CoV-2 en fonction de la phylogénie.
- A [2019-12-30] (second haplotype découvert – après « B » – ; la racine de la pandémie se situe dans cette lignée A).
- A.23
- A.23.1 [2020-10-21]
- B [2019-12-24] (premier haplotype découvert).
- B.1 [2020-01-24]
- B.1.1 [2020-02-16]
- B.1.1.1
- C.1, alias de B.1.1.1.1
- C.1.2, [2021-05-11], alias de B.1.1.1.1.2
- C.36, alias de B.1.1.1.36
- C.36.3, alias de B.1.1.1.36.3
- C.36.3.1 [2021-03-27], alias de B.1.1.1.36.3.1
- C.37 [2020-11-08], alias de B.1.1.1.37 (Lambda)
- B.1.1.7 [2020-02-07] (Alpha)
- Q.* [2020-07-11] à [2021-03-02], alias de B.1.1.7.*
- B.1.1.28
- P.1 [2020-04-07], alias de B.1.1.28.1 (Gamma)
- P.1.1 [2021-01-07], alias de B.1.1.28.1.1
- P.1.2 [2021-01-14], alias de B.1.1.28.1.2
- P.1.4 [2021-03-19], alias de B.1.1.28.1.4
- P.1.6 [2021-03-26], alias de B.1.1.28.1.6
- P.1.7 [2021-03-10], alias de B.1.1.28.1.7
- P.2 [2020-04-13], alias de B.1.1.28.2 (Zêta)
- P.3 [2021-01-08], alias de B.1.1.28.3 (Thêta)
- P.1 [2020-04-07], alias de B.1.1.28.1 (Gamma)
- B.1.1.316
- R.1 [2020-01-14], alias de B.1.1.316.1
- R.2 [2020-12-22], alias de B.1.1.316.2
- B.1.1.318 [2021-01-07]
- B.1.1.370
- AT.1 [2021-01-18], alias de B.1.1.370.1
- B.1.1.482
- AV.1 [2021-02-18], alias de B.1.1.482.1
- B.1.1.519 [2020-07-22]
- B.1.1.529 [2021-11-08] (Omicron)
- BA.1 [2021-01-12], alias de B.1.1.529.1 (Omicron)
- XD [2022-01-03], recombinant de Delta – lignée AY.4 (B.1.617.2.4) et BA.1 — voir également AY.4 (Delta)
- XE [2022-01-19], recombinant de BA.1 et BA.2 — voir également BA.2 (Omicron)
- BA.2 [2021-11-17], alias de B.1.1.529.2 (Omicron)
- XE [2022-01-19], recombinant de BA.1 et BA.2 — voir également BA.1 (Omicron)
- BA.3 [2021-11-25], alias de B.1.1.529.3 (Omicron)
- BA.4 [2022-01-10], alias de B.1.1.529.4 (Omicron)
- BA.5 [2022-01-06], alias de B.1.1.529.5 (Omicron)
- BA.1 [2021-01-12], alias de B.1.1.529.1 (Omicron)
- B.1.1.1
- B.1.177 [2020-02-14]
- B.1.214
- B.1.214.2 [2020-11-22]
- B.1.351 [2020-05-09] (Bêta)
- B.1.351.1 [2021-01-19]
- B.1.351.2 [2020-12-06]
- B.1.351.3 [2021-01-12]
- B.1.427 [2020-04-11]
- B.1.429 [2020-03-11]
- B.1.466
- B.1.466.2 [2020-08-06]
- B.1.525 [2020-12-11] (Êta)
- B.1.526 [2020-04-21] (Iota)
- B.1.617
- B.1.617.1 [2020-12-01] (Kappa)
- B.1.617.2 [2020-09-07] (Delta)
- AY.1 [2021-04-05], alias de B.1.617.2.1
- AY.2 [2021-03-12], alias de B.1.617.2.2
- AY.3 [2021-04-23], alias de B.1.617.2.3
- AY.3.1 [2020-11-20], alias de B.1.617.2.3.1
- AY.4 [2020-05-11], alias de B.1.617.2.4
- XD [2022-01-03], recombinant de Delta – lignée AY.4 (B.1.617.2.4) et BA.1 — voir également BA.1 (Omicron)
- AY.5 ... AY.12 [2020-07-13](AY.10) ... [2020-11-15](AY.5), alias de B.1.617.2.5 ... B.1.617.2.12
- AY.13 ... AY.25 [2020-07-21](AY.25) ... [2021-05-30](AY.23.1), alias de B.1.617.2.13 ... B.1.617.2.25
- B.1.619 [2020-03-10]
- B.1.619.1 [2021-02-06]
- B.1.620 [2021-02-06]
- B.1.621 [2021-01-11] (Mu)
- B.1.621.1 [2021-03-18]
- B.1.1 [2020-02-16]
- A.23
Public Health England
Lors de la détection d'un variant dans le Kent au sud-est de Londres, l'agence exécutive gouvernementale britannique Public Health England (PHE) a défini une nomenclature.
En septembre 2020, PHE a détecté un variant qui s'est révélé digne d'intérêt en , et l'a appelé, le , « first Variant Under Investigation in December 2020 » (VUI-202012/01, pour Variant Under Investigation, year 2020, month 12, variant 01). Ce variant a été rebaptisé « variant préoccupant » (Variant of Concern, VOC) le , soit alors « VOC-202012/01 ».
Mi-, PHE a modifié ses règles de nommage en : [VOC/VUI] – [deux derniers chiffres de l'année] – [trois caractères du mois] – [numéro de variant dans le mois sur deux digits]. Depuis lors, « VOC-202012/01 » est connu comme « VOC-20DEC-01 ».
Public Health England a également été chargé du séquencement du variant dit « sud-africain », en . Ce variant a alors reçu le nom de « VOC-202012/02 ». Il est depuis nommé « VOC-20DEC-02 » (synonymes : 501Y.V2, B.1.351).
Pour les variants dits « brésiliens », Public Health England a effectué le séquençage d'un variant en , depuis nommé « VUI-21JAN-01 » (synonymes : Zêta, ou P.2 – descendant de B.1.1.28), et d'un variant détecté au Japon, sur des voyageurs en provenance du Brésil, depuis nommé « VOC-21JAN-02 » (synonymes : Gamma, ou P.1 – descendant de B.1.1.28).
Noms et équivalences
Depuis le , l'OMS a défini la règle de nommage des variants, et catégorisé ces variants (VOC/VOI/VUM). Dans les tableaux suivants, c'est cette qualification qui est retenue.
Cependant, le nommage des variants, et leur catégorisation évolue dans le temps. Ainsi, l'état est ici indiqué pour la dernière publication de l'OMS connue : au .
- :
- • définition de la règle de nommage ;
- • publication des VOC « Alpha », « Bêta », « Gamma » et « Delta » ;
- • publication des VOI « Epsilon », « Zêta », « Êta », « Thêta », « Iota », « Kappa » ;
- :
- • publication du VOI « Lambda » ;
- :
- • classification de nouvelles lignées, pour les variants Bêta et Gamma :
- outre B.1.351, Bêta est maintenant défini comme B.1.351 + B.1.351.2 + B.1.351.3
- outre P.1, Gamma est maintenant défini comme P.1 + P.1.1 + P.1.2
- • surveillances additionnelles introduites pour les variants Alpha et Gamma :
- pour Alpha, surveillance des mutations S:484K et S:452R ;
- pour Gamma, surveillance de la mutation S:417N ;
- :
- • surveillances additionnelles introduites pour les variants Bêta et Delta :
- pour Bêta, surveillance de la mutation S:L18F ;
- pour Delta, surveillance de la mutation S:417N ;
- • les variants Epsilon, Zêta, Thêta sont requalifiés de VOI à VUM ;
- • les variants VUM sont publiés :
- B.1.427/B.1.429 (ex-
Epsilon) - P.2 (ex-
Zêta) - P.3 (ex-
Thêta) - R.1 & R.2
- B.1.466.2
- B.1.621
- AV.1
- B.1.1.318
- B.1.1.519
- AT.1
- C.36.3 + C.36.3.1
- B.1.214.2
- B.1.427/B.1.429 (ex-
- :
- • classification d'une nouvelle lignée intégrée au variant Delta :
- outre B.1.617.2 + AY.1 + AY.2, Delta est maintenant défini comme B.1.617.2 + AY.1 + AY.2 + AY.3
- • les variants B.1.1.523, B.1.619 et B.1.620 sont ajoutés à la liste des VUM.
- :
- • dans la liste des VUM, retrait de AV.1 & AT.1, qui ne sont maintenant plus suivis ;
- :
- • classification de nouvelles lignées, pour des noms de variants existants :
- outre P.1 + P.1.1 + P.1.2, Gamma est maintenant défini comme P.1 + P.1.1 + P.1.2 + P.1.4 + P.1.6 + P.1.7
- outre B.1.617.2 + AY.1 + AY.2 + AY.2, Delta est maintenant défini comme B.1.617.2 + AY.1 + AY.2 + AY.3 + AY.3.1
- • dans la liste des VUM :
- retrait de P.2 (ex-
Zêta), P.3 (ex-Thêta) et R.2, qui ne sont maintenant plus suivis ; - la surveillance de B.1.619 est étendue à celle de B.1.619 + B.1.619.1 ;
- la surveillance de B.1.621 est étendue à celle de B.1.621 + B.1.621.1 ;
- retrait de P.2 (ex-
- :
- • classification de nouvelles lignées, pour des noms de variants existants :
- outre B.1.1.7, Alpha est maintenant défini comme B.1.1.7 + Q.* (c.-à-d. B.1.1.7.* — « * » signifiant « n'importe quel suffixe » ) ;
- outre B.1.351, Bêta est maintenant défini comme B.1.351 + B.1.351.2 + B.1.351.3 ;
- Gamma, auparavant défini comme P.1 + P.1.1 + P.1.2 + P.1.4 + P.1.6 + P.1.7, est maintenant réduit à P.1 + P.1.4 + P.1.6 + P.1.7 ;
- Delta, auparavant défini comme B.1.617.2 + AY.1 + AY.2 + AY.3 + AY.3.1, est maintenant défini comme B.1.617.2 + AY.* (c.-à-d. B.1.617.2.*) — « * » signifiant « n'importe quel suffixe » ) ;
- :
- • classification de nouvelles lignées, pour des noms de variants existants :
- Gamma, auparavant défini comme P.1 + P.1.4 + P.1.6 + P.1.7, est maintenant réduit à P.1 ;
- • publication du VOI « Mu » (synonyme de B.1.621) ;
- :
- • dans la liste des VUM, ajout de C.1.2 ;
- :
- • les variants Êta, Iota, Kappa ont été requalifiés de VOI à VUM le ;
Variants préoccupants (VOC)
Les variants Alpha, Bêta, Gamma, Delta et Omicron sont classés par l'OMS comme préoccupants.
| Nom officiel de l'OMS |
Nextstrain | Lignées Pango | GISAID | Public Health England (PHE) |
Date et lieu de première détection |
Mutations clés | Mutations en cours d’acquisition ? | |
|---|---|---|---|---|---|---|---|---|
|
Alpha VOC : 2020-12-18 + au 2021-07-01, surveillance de S:484K et S:452R |
20I/501Y.V1 | B.1.1.7 | GRY (précédemment GR/501Y.V1 |
VOC-20DEC-01 précédemment: VOC-202012/01 (first VOC of december 2020) |
Royaume-Uni |
ORF1a : T1001I • A1708D • I2230T • Δ3675S/3677F ; ORF1b : P314L ; S : Δ69/70 • Δ144Y • N501Y • A570D • D614G • P681H • H655Y • T716I • S982A • D1118H ; ORF8 : Q27* • R52I • Y73C ; N : D3L • R203K • G204R • S235F  Cartographie des mutations de codons du génome de SARS-CoV-2. |
S : S477R? • E484K • F490S • D614-(LYQDVNC) | |
|
Bêta VOC : 2020-12-18 + au 2021-07-06, surveillance de S:L18F |
20H/501Y.V2 | B.1.351 + au 2021-07-01 B.1.351.2 B.1.351.3 |
GH/501Y.V2 | VOC-20DEC-02 précédemment : VOC-202012/02 (second VOC of december 2020) |
Afrique du Sud |
ORF1a : T265I • K1655N • K3353R • Δ3675S/3677F ; ORF1b : P314L ; S : L18F • D80A • D215G • Δ241/243 • K417N • E484K • N501Y • D614G • A701V ; ORF3a : Q57H • S171L ; E : P71L ; N : T205I  Cartographie des mutations de codons du génome de SARS-CoV-2. |
S : V483F? | |
|
Gamma VOC : 2021-01-11 + au 2021-07-01, surveillance de S:681H |
20J/501Y.V3 |
P.1(alias B.1.1.28.1) + au 2021-07-01 P.1.1 P.1.2 |
GR/501Y.V3 | VOC-21JAN-02 précédemment : VOC-202101/02 |
Brésil / Japon |
ORF1a : S1188L • K1795Q • Δ3675S/3677F ; ORF1b : P314L • E1264D ; S : L18F • T20N • P26S • D138Y • R190S • K417N/T • E484K • N501Y • D614G • H655Y • T1027I • V1176F ; ORF3a : S253P ; ORF8 : E92K ; N : P80R • R203K • G204R ; ORF9b : Q77E  Cartographie des mutations de codons du génome de SARS-CoV-2. |
S : T470N? | |
|
Delta VOI : 2021-04-04 VOC : 2021-05-11 + au 2021-07-06, surveillance de S:417N |
21A ou 21A/S:478K |
B.1.617.2 + au 2021-07-01 AY.1 AY.2 |
G/478K.V1 | VOC-21APR-02 |
Inde |
ORF1a : T3255I ; ORF1b : P314L • G662S • P1000L ; S : T19R • G142D • Δ156/157 • R158G • L452R • T478K • D614G • P681R • D950N ; ORF3a : S26L ; M : I82T ; ORF7a : V82A • T120I ; ORF8 : Δ119D/120F ; N : D63G • R203M • D377Y ; ORF9b : T60A |
S : A475S? • S477I ? • P479L ? • P479S ? • D614-(LYQDVNC) | |
|
Omicron dates à préciser |
lignée d'origine B.1.1.529 (alias BA.1) |
21M | B.1.1.529 + au 2021-12-07 BA.1 BA.2 |
GR/484A |
Afrique du Sud |
|
||
| (sous-lignée BA.1) | 21K | BA.1 | GR/484A |
 {{{1}}} |
||||
| (sous-lignée BA.2) | 21L (22C pour BA.2.12.1) |
BA.2 | GR/484A |
 {{{1}}} |
||||
| (sous-lignée BA.3) | — aucun clade défini — | BA.3 | GR/484A |
 Cartographie des mutations de codons du génome de SARS-CoV-2. |
||||
| (sous-lignée BA.4) | 22A | BA.4 | GR/484A |
 Cartographie des mutations de codons du génome de SARS-CoV-2. |
||||
| (sous-lignée BA.5) | 22B | BA.5 | GR/484A |
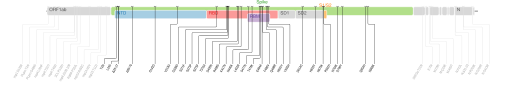 Cartographie des mutations de codons du génome de SARS-CoV-2. |
||||
Sources : OMS, PANGOlin, Public Health England, Santé Publique France.
Notes VOC du tableau :
Variants d'intérêt (VOI)
| Nom officiel de l'OMS |
Nextstrain | Lignées Pango | GISAID | Public Health England (PHE) |
Date et lieu de première détection |
Mutations clés | Mutations en cours d’acquisition ? |
|---|---|---|---|---|---|---|---|
| qualification OMS : ex- VOI : 2021-03-05 requalifié VUM le 2021-09-20 | |||||||
| qualification OMS : ex- VOI : 2021-03-17 requalifié VUM le 2021-07-06 exclu de la liste des VUM le 2021-08-13, n'est alors plus suivi | |||||||
| qualification OMS : ex- VOI : 2021-03-17 requalifié VUM le 2021-09-20 | |||||||
| qualification OMS : ex- VOI : 2021-03-24 requalifié VUM le 2021-07-06 exclu de la liste des VUM le 2021-08-13, n'est alors plus suivi | |||||||
| qualification OMS : ex- VOI : 2021-03-24 requalifié VUM le 2021-09-20 | |||||||
| qualification OMS : ex- VOI : 2021-04-04 requalifié VUM le 2021-09-20 | |||||||
|
Lambda VOI : 2021-06-14 |
|
C.37 | GR/452Q.V1 | VUI-21JUN-01 |
Pérou |
ORF1a : T1246I • P2287S • F2387V • L3201P • T3255I • G3278S • Δ3675S/3677F • ORF1b : P314L • S : G75V • T76I • Δ246R/252G • L452Q • F490S • D614G • T859N • N : P13L • R203K G204R • G214C 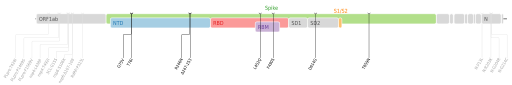 Cartographie des mutations de codons du génome de SARS-CoV-2. |
|
|
Mu VOI : 2021-08-30 |
21H | B.1.621 |
Colombie |
ORF1a : T1055A • T1538I • T3255I • Q3729R • ORF1b : P314L • P1342S • S : T95I • Y144S • Y145N • R346K • E484K • N501Y • D614G • P681H • D950N • ORF3a : Q57H • ORF8 : T11K • P38S • S67F • N : T205I  Cartographie des mutations de codons du génome de SARS-CoV-2. |
|||
Sources : OMS, PANGOlin, Public Health England, Santé Publique France.
Notes VOI du tableau :
Variants en cours d’évaluation (VUM)
| Nom officiel de l'OMS |
Nextstrain | Lignées Pango | GISAID | Public Health England (PHE) |
Date et lieu de première détection |
Mutations clés | Mutations en cours d’acquisition ? |
|---|---|---|---|---|---|---|---|
| ex- VOI : 2021-03-05 requalifié VUM le 2021-07-06 |
20C/S:452R | B.1.427/B.1.429 | GH/452R.V1 |
États-Unis |
ORF1a : T265I • I4205V • ORF1b : P314L • D1183Y • S : S13I • W152C • L452R • D614G • ORF3a : Q57H • ORF8 : V100L • N : T205I • M234I  Cartographie des mutations de codons du génome de SARS-CoV-2. |
||
| qualification OMS : ex- VOI : 2021-03-17 requalifié VUM le 2021-07-06 exclu de la liste des VUM le 2021-08-13 : n'est alors plus suivi | |||||||
| ex- VOI : 2021-03-17 |
20C | B.1.525 | G/484K.V3 | VUI-21FEB-03 |
Royaume-Uni / Nigéria |
ORF1a : T2007I • Δ3675S/3677F • ORF1b : P314F • S : Q52R • A67V •Δ69H/70V • Δ144Y • E484K • D614G • Q677H • F888L • E : L21F • M : I82T • ORF6 : Δ2F • N : S2M • D3Y • A12G • T205I • ORF9b : H9D 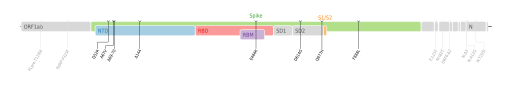 Cartographie des mutations de codons du génome de SARS-CoV-2. |
|
| qualification OMS : ex- VOI : 2021-03-24 requalifié VUM le 2021-07-06 exclu de la liste des VUM le 2021-08-13 : n'est alors plus suivi | |||||||
| ex- VOI : 2021-03-24 (S477N ou E484K) |
21F | B.1.526 | GH |
New York City |
ORF1a : T265I • L3201P • Δ3675S/3677F • ORF1b : P314L • Q1011H • S : L5F • T95I • D253G • S477N ou E484K (au choix) • D614G • ORF3a : P42L • Q57H • ORF8 : T11I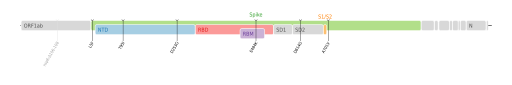 Cartographie des mutations de codons du génome de SARS-CoV-2. |
||
| ex- VOI : 2021-04-04 |
21B | B.1.617.1 | G/452R.V3 | VUI-21APR-01 |
Inde |
ORF1a : T1567I • T3646A • ORF1b : P314L • G1129C • M1352I • K2310R • S2312A • S : L452R • E484Q • D614G • P681R • Q1071H • ORF3a : S26L • M : I82S • ORF7a : V82A • N : R203M • D377Y  Cartographie des mutations de codons du génome de SARS-CoV-2. |
|
| (20A) | B.1.620 |
Afrique |
ORF1a : T403I • V1991I • Δ3675S/3677F • ORF1b : P314L • A1215S • S : P26S • Δ69H/70V • V126A • Δ144Y • Δ241/243 • H245Y • S477N • E484K • D614G • P681H • T1027I • D1118H • ORF7b : Δ144L • N : A220V • ORF9b : I5T | ||||
| (20D) | C.36.3 |
Asie |
ORF1a : E102K • T1246I • D1639N • D2980N • D3222N • G3278S • S3687L • L3691S • T4090I • ORF1b : P314L • D1028Y • S : S12F • W152R • D614G • Q677H • M : I82T • ORF7b : A43S • N : R203K • G204R • G212V | S : Q474H ? | |||
| (20B) | B.1.1.318 |
Afrique |
ORF1a : K2511N • T2936I • A3209V • Δ3675S/3677F • ORF1b : P314L • V2371M • S : T95I • Δ144Y • E484K • D614G • P681H • D796H • M : I82T • ORF7b : *44X • ORF8 : Δ1M/2K • F3X • E106X • *122X • N : R203K • G204R • Δ208A • R209G | ||||
| (20B) | AT.1 |
Russie |
ORF1a : L39K • S376L • V1006F • T1093A • T2247N • T3255I • Q3729R • S4119T • ORF1b : A190S • P314L • T1173N • V1905L • A2431V • S : P9L • Δ136C/144Y • D215G • H245P • E484K • D614G • E780K • ORF3a : L95M • M : L16I • N : R203K • G204R | ||||
| (20B) | B.1.1.519 |
Mexique |
ORF1a : P959S • I3618V • T4175I • ORF1b : P314L • S : T478K • D614G • P681H • T732A • N : R203K • G204R | S : D614-(LYQDVNC) | |||
Sources : OMS, PANGOlin, Public Health England, Santé Publique France.
Notes VUM du tableau :
Variants non-suivis
Ce sont les variants, précédemment classifiés comme VOI, qui ont été successivement requalifiés de VOI à VUM, puis de VUM à non-suivi (noté ici NS)
| Nom officiel de l'OMS |
Nextstrain | Lignées Pango | GISAID | Public Health England (PHE) |
Date et lieu de première détection |
Mutations clés | Mutations en cours d’acquisition ? |
|---|---|---|---|---|---|---|---|
| ex- VOI : 2021-03-17 requalifié VUM le 2021-07-06 exclu de la liste des VUM le 2021-08-13, n'est alors plus suivi |
20J | P.2 (alias B.1.1.28.2) | GR | VUI-21JAN-01 |
Brésil |
ORF1a : L3468V • L3930F • ORF1b : P314L • S : E484K • D614G • V1176F • N : A119S • R203K • G204R • M234I 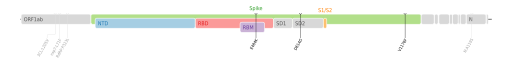 Cartographie des mutations de codons du génome de SARS-CoV-2. |
|
| ex- VOI : 2021-03-24 requalifié VUM le 2021-07-06 exclu de la liste des VUM le 2021-08-13, n'est alors plus suivi |
21E | P.3 | GR | VUI-21MAR-02 |
Philippines |
ORF1a : D1554G • L3201P • D3681E • L3930F • ORF1b : P314L • A1291V • S : Δ141L/143V • E484K • N501Y • D614G • P681H • E1092K • H1101Y • V1176F • ORF8 : K2Q • N : R203K • G204R 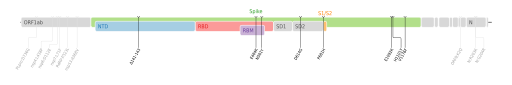 Cartographie des mutations de codons du génome de SARS-CoV-2. |
Sources : OMS, PANGOlin, Public Health England, Santé Publique France.
Notes NS du tableau :
Références croisées variants/mutations
Dans le cas d'une infection par le SARS-CoV-2, il est suggéré que seuls les anticorps ciblant la protéine S du SARS-CoV-2 sont neutralisants. Les anticorps contre la protéine S ne ciblent que quelques fragments spécifiques :
- les anti-RBD représentent entre 65 et 77 % des anti-S. Les anti-RBD opsonisent le RBM localisé entre les codons 437 et 508 de la protéine S. Au sein du RBM, trois segments contiennent des épitopes reconnus par les anticorps : la crête (receptor-binding ridge - RBR) localisée aux codons 417, 455-456 et 470–490, et deux boucles (loop) localisées aux codons 443-452 (L1) et 494-501(L2). Le RBR est particulièrement important car il entre en contact direct avec le récepteur cellulaire ACE2 ;
- les anti-NTD représentent entre 6 et 20 % des anti-S. La plupart des anticorps dirigés contre le NTD reconnaissent une « boucle » (loop en anglais), qui est généralement la boucle N1 (localisée aux codons 14 à 26), la boucle N3 (codons 141 à 156) ou la boucle N5 (codons 246 à 260).
Un variant qui cumulerait un grand nombre de mutations sur ces codons (14-26 ; 141-156 ; 246-260 ; 437-508) serait susceptible d'échapper totalement aux anticorps neutralisants induits par les vaccins.
| Gène | Codon mutant | Localisation | Alpha (B.1.1.7) | Beta (B.1.351) | Iota (B.1.526) | Kappa (B.1.617.1) | Delta (B.1.617.2) | Eta (B.1.525) | Lambda (C.37) | Theta (P.3) | Gamma (P.1) | Mu (B.1.621) | C.36.3 | B.1.1.318 | B.1.620 | AT.1 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Origine - 1er séquençage | Angleterre - 20 septembre 2020 | Afrique du Sud - 08 oct 2020 | New York - 23 novembre 2020 | Inde - 1er décembre 2020 | Inde - 10 décembre 2020 | Nigéria - 11 décembre 2020 | Andes (Chili & Pérou) - 22 décembre 2020 | Philippines - 16 janvier 2021 | Brésil - 16 janvier 2021 | Colombie - 8 mars 2021 | Asie – 26 janvier 2021 | Afrique – 7 janvier 2021 | Afrique – 6 février 2021 | Russie - 18 janvier 2021 | ||
| Nombre de mutations dont : | 29 | 23 | 19 | 20 | 23 | 25 | 31 | 26 | 26 | 21 | 27 | 26 | 26 | |||
| ORF1a | L3201P | nsp4 (L438P) | 1 | 1 | 1 | |||||||||||
| ORF1a | T3255I | nsp5 | 1 | 1 | 1 | 1 | ||||||||||
| ORF1a | S3675- | nsp6 (S106-) | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | ||||||
| ORF1a | G3676- | nsp6 (G107-) | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | ||||||
| ORF1a | F3677- | nsp6 (F108-) | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | ||||||
| ORF1b | P314L / P314F | nsp12 (RDPR : P323L) | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| S | H69- | NTD (loop N2) | 1 | 1 | 1 | 1 | ||||||||||
| S | V70- | NTD (loop N2) | 1 | 1 | 1 | 1 | ||||||||||
| S | T95I | NTD | 1 | 1 | 1 | 1 | ||||||||||
| S | F140- | NTD (loop N3) | 1+ | 1+ | 1 | |||||||||||
| S | G142- / G142D | NTD (loop N3) | 1 | 1 | 1 | |||||||||||
| S | Y144- / Y144S | NTD (loop N3) | 1 | 1 | 1 | 1 | 1 | 1 | ||||||||
| S | L242- | NTD | 1 | 1 | 1 | |||||||||||
| S | A243- | NTD | 1 | 1 | 1 | |||||||||||
| S | Y248- | NTD (loop N5) | 1 | 1+ | ||||||||||||
| S | L249- | NTD (loop N5) | 1 | 1+ | ||||||||||||
| S | K417N / K417T | RBD (RBM-RBR) | 1 | 1+ | 1 | |||||||||||
| S | L452R / L452Q | RBD (RBM-L1) | 1 | 1 | 1 | 1 | ||||||||||
| S | S477N | RBD (RBM-RBR) | 1re lignée | 1 | ||||||||||||
| S | T478K | RBD (RBM-RBR) | 1 | |||||||||||||
| S | E484K / E484Q | RBD (RBM-RBR) | 1 | 2e lignée | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | ||||
| S | F490S | RBD (RBM-RBR) | 1 | |||||||||||||
| S | N501Y | RBD (RBM-L2) | 1 | 1 | 1 | 1 | 1 | |||||||||
| S | D614G | SD2 (« LYQDVNC ») - ADE | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| S | P681H / P681R | SD2 (NSPRRAR') - CendR/neuropiline 1 & furine | 1 | 1 | 1 | 1 | 1 | 1 | 1 | |||||||
| ORF3a | Q57H | ORF3c & 3d | 1 | 1 | 1 | |||||||||||
| ORF3a | V163L | ORF3b (codon 22) | 1+ | |||||||||||||
| M | I82T / I82S | 1 | 1 | 1 | 1 | 1 | ||||||||||
| N | R203K / R203M | ORF9c | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | |||||
| N | G204R | ORF9c | 1 | 1 | 1 | 1 | 1 | 1 | 1 | |||||||
| N | T205I | ORF9c | 1 | 1 | 1 |
Variants
Variants préoccupants
Alpha (B.1.1.7)

Détecté pour la première fois en lors de la pandémie de Covid-19 au Royaume-Uni à partir d'un échantillon prélevé le mois précédent, le variant alpha, ou VOC-202012/01 (first Variant Of Concern in December 2020), auparavant connu sous le nom « VUI-1202012/01 » (first Variant Under Investigation in December 2020), est également nommé « B.1.1.7 » (souche, ou lignée, B.1.1.7) ou encore « 20B/501Y.V1 ». Depuis lors, sa probabilité de prévalence a doublé tous les 6,5 jours[réf. nécessaire], l'intervalle générationnel présumé. Son apparition est corrélée à une augmentation significative du taux d'infection au Covid-19 au Royaume-Uni, associée en partie à la mutation N501Y, qui modifie la façon dont le virus s'attache aux cellules par la glycoprotéine S.
Il a connu une croissance exponentielle à partir de début pour devenir largement majoritaire. Il serait de 50 à 75 % plus contagieux que les souches habituelles, y compris chez les enfants, mais n'entraînerait pas plus de formes graves. Ce variant se propage alors dans le monde.
Bêta (B.1.351)
Le 18 décembre 2020, le variant appelé 501.V2, 20C/501Y.V2 ou lignée B.1.351 est détecté pour la première fois en Afrique du Sud et signalé par le département de la santé du pays. Les chercheurs ont signalé que la prévalence du variant était plus élevée chez les jeunes sans problèmes de santé sous-jacents et que, par comparaison avec d'autres variants, il entraînait plus fréquemment une maladie grave dans ces cas. Le département de la santé sud-africain a également indiqué que le variant pourrait être à l'origine de la deuxième vague de l'épidémie de COVID-19 dans le pays en raison de sa propagation à un rythme plus soutenu que les autres variants du virus.
Les scientifiques ont également noté que le variant contenait plusieurs mutations lui permettant de se fixer plus facilement aux cellules humaines en raison des trois mutations suivantes dans le domaine de liaison au récepteur (RBD – receptor binding domain) dans la glycoprotéine de pointe du virus : N501Y, K417N et E484K. La mutation E484K pourrait altérer l'efficacité des vaccins en réduisant le pouvoir neutralisant des anticorps. Il entraînerait alors aussi des risques de réinfection.
Gamma (P.1)
Le variant Gamma du SARS-CoV-2 a été détecté la 1re fois à Tokyo le par l'Institut national des maladies infectieuses (NIID). Ce nouveau variant a été trouvé chez quatre personnes arrivées le à Tokyo depuis l'État d'Amazonas au Brésil. L'Institut Oswaldo-Cruz indique que le variant aurait circulé dans la forêt amazonienne. Ce variant a 12 mutations dans sa protéine de pointe, y compris N501Y et E484K. Une prépublication d'un article de Carolina M Voloch et al. suppose que le variant serait apparu pour la première fois en , avant d'être détecté par eux-mêmes en .
Le variant Gamma est responsable d'une seconde vague extrêmement intense dans l'État de l'Amazonas et plus particulièrement dans la ville de Manaus, alors même que la ville avait atteint le seuil d'immunité collective en avec 76 % de la population déjà exposée au Covid-19. Il a été spéculé que l’immunité acquise avec d'anciennes souches du virus ne permettait pas d’être protégé face à ce variant. Le variant Gamma circule au côté du variant Zêta (P.2) détecté à Rio de Janeiro. Les deux variants sont issus du variant B.1.1.28 qui aurait circulé au Brésil depuis .
Delta (B.1.617.2)
Détecté le près de Nagpur en Inde, le variant B.1.617 présente 15 mutations dont deux dans le domaine de liaison RBD de la protéine spiculaire :
- L452R, détectée chez les variants B.1.427/B.1.429 (Californie) ;
- E484Q, affectant le même acide aminé que la mutation E484K des variants sud-africains et brésiliens.
Ces deux dernières mutations sont préoccupantes car elles pourraient lui permettre d'échapper aux anticorps (post-infection ou vaccinaux). Elles lui valent le qualificatif (abusif) de « double mutant ».
En avril 2021, il s'était propagé à au moins huit pays. Des rapports de terrain le décrivent comme plus contagieux. Ses symptômes seraient différents (on parle de saignements de nez).
Santé publique France écrit le : « Ce variant a été détecté sporadiquement en Angleterre, Allemagne, au Canada et à Singapour. Deux cas ont été détectés en Guadeloupe. »
Fin avril, il s'avère que ce variant circule aux Fidji. Il y a été apporté par deux patients rapatriés d'Inde. Un soldat en centre de quarantaine a alors été contaminé en touchant leurs bagages. Il a ensuite contaminé une femme de ménage, qui n'a pas informé les autorités de signes cliniques de sa maladie et s'est déplacée librement en dehors du centre de quarantaine, contaminant de nombreuses autres personnes.
Le 29 avril 2021, l'agence régionale de santé de Nouvelle-Aquitaine annonce que le variant indien a été détecté pour la première fois en France métropolitaine, dans le département de Lot-et-Garonne, sur un patient revenu d'Inde. Le lendemain, la Direction générale de la Santé révèle que deux nouveaux cas ont été détectés dans les Bouches-du-Rhône et que d'autres cas suspects sont en cours d'investigation dans d'autres régions, concernant toujours des personnes venues d'Inde.
Le 10 mai 2021, ce variant a été classé comme « préoccupant » par l'OMS.
Le 11 mai 2021, ce variant — dit indien — est présent dans sept des treize régions métropolitaines de France.
Le 14 mai 2021, le variant B.1.617 entre à São Luís, Maranhão, Brésil, par bateau, en provenance de Malaisie via Le Cap.
Fin mai 2021, les sous-lignées B.1.617.1 et B.1.617.2 sont nommées respectivement Kappa et Delta. Le nombre de contaminations repart à la hausse au Royaume-Uni en raison du variant Delta, car deux doses sont nécessaires pour protéger du variant delta.
En juin 2021, le variant Delta représente 75 % des contaminations au Royaume-Uni et continue de se propager dans des départements du sud-ouest de la France où l'épidémie a un taux de reproduction de nouveau supérieur à 1, alors que les autres départements de la métropole sont encore en phase de baisse de l'incidence.
Delta plus
Parallèlement, un sous-variant du variant Delta est apparu au Népal avec la mutation K417N déjà présente dans les variants Bêta et Gamma. Cette mutation pourrait favoriser l'échappement aux anticorps et donner un risque accru de réinfection. Le variant, appelé « Delta avec K417N » par Public Health England, comprend deux clades correspondant aux lignées Pango AY.1 et AY.2. Il a été surnommé « Delta plus » (pour « Delta, plus K417N »). Le , le ministère indien de la santé et du bien-être familial a classé le variant Delta plus comme préoccupant après le signalement de seulement 22 cas, ce qui serait prématuré d'après d'éminents virologues. Début , ce variant est présent dans plusieurs pays mais sa diffusion n'est significative qu'au Népal avec 7 % des séquencements.
Autres lignées Delta
Selon Pango, ses règles de classification de nouvelles lignées et les classifications AY.1 à AY.3 déjà publiées, les sous-lignées du variant Delta (c.-à-d. B.1.617.2 alias AY) comprennent maintenant, au , les lignées AY.4 à AY.12. Ces lignées appartiennent au variant Delta, et sont nommées pour faciliter les recherches scientifiques (meilleure granularité génomique, meilleure détermination de répartition géographique).
Au , Pango a émis une mise à jour pour les variants AY.4 à AY.12, introduisant également les lignées AY.13 à AY.25, lignées appartenant toujours au variant Delta.
Omicron (B.1.1.529)
Découvert en Afrique du Sud en novembre 2021, le variant B.1.1.529 désigné par l'OMS sous le nom de variant Omicron est un variant désigné comme préoccupant par l'OMS, en effet celui-ci contiendrait un nombre extrêmement élevé de mutations : une trentaine environ sur la protéine spike contre deux pour le variant Delta.
Sous-variant BA.2
Un sous-variant du variant Omicron, nommé BA.2 (alias B.1.1.529.2), détecté en Inde, est devenu dominant en janvier 2022 au Danemark.
De même, les variants BA.1 et BA.3, sous-variants de B.1.1.529, sont également qualifiés de variants Omicron.
Recombinant « Deltacron » XD
Le 11 février 2022, l'Agence de sécurité sanitaire du Royaume-Uni (UKHSA) a placé sous surveillance une recombinaison Delta × Omicron suspectée chez un patient infecté simultanément par les deux variants Delta et Omicron.
Le variant « Deltacron » XD a été identifié pour la première fois en janvier 2022 à Chypre. Il est proche du Delta (souche AY.4) avec un apport Omicron (souche BA.1) sur le gène S (codant la protéine spiculaire). Son développement reste marginal comparé au BA.2.
Recombinant XE
Le variant XE est un recombinant BA.1 (Omicron original B.1.1.529.1) et BA.2 (apparenté Omicron B.1.1.529.2). Détecté en au Royaume-Uni, son développement reste également marginal.
Sous-variants BA.4 et BA.5
Les variants BA.4 et BA.5 sont des variants issus de la famille Omicron, classés comme variants d’intérêt par l'OMS le puis relevés au rang de variants préoccupants le . Leur ancêtre commun serait apparu à la mi-novembre 2021 avant qu'ils soient détectés en Afrique du Sud mi-décembre 2021 et début janvier 2022. Ils ont la même protéine spiculaire, proches de celle du variant BA.2 mais s'en distinguent par la délétion Δ69-70 — dans le domaine amino-terminal (NTD) — et les mutations L452R et F486V — affectant le domaine de liaison au récepteur (RBD). Ces variants sont responsables de la flambée épidémique du printemps et de l'été 2022 en Europe occidentale. Un sous-variant BA.5.3.1 représenterait 80 % des cas de BA.5 en Allemagne.
Futurs variants
En juillet 2022, de nouveaux sous-variants issus de la famille Omicron sont placés sous surveillance : le BA.2.75 « Centaure » et le BA.5.3.1 « Bad NED ». Ils semblent indiquer que le SARS-CoV-2 continue à évoluer tout en restant dans le cadre des lignées Omicron, un terrain qui lui offre aujourd'hui les meilleures perspectives pour continuer à gagner en efficacité.
Octobre 2022 voit notamment l'émergence du sous-variant recombinant XBB (BJ.1 × BA.2.75).
Aux États-Unis, en novembre 2022, le nouveau variant BN.1 avec la mutation R346T qui échappe à l'immunité et "au médicament utilisé par les personnes au système immunitaire affaibli", pourrait "doubler son nombre de contaminations tous les quinze jours".
Variants d'intérêt
Êta (B.1.525)
Le variant Êta ou B.1.525, encore appelé VUI-21FEB-03 (auparavant VUI-202102/03) par Public Health England et auparavant connu comme UK1188, 21D ou 20A/S:484K, ne porte pas la même mutation N501Y que les variants Alpha, Beta et Gamma mais porte la même mutation E484K que les variants Beta, Gamma et Zéta ainsi que la même délétion ΔH69/ΔV70 que le variant Alpha, N439K (B.1.141 et B.1.258) et Y453F variant (Cluster 5). Êta diffère de tous les autres variants par la présence simultanée des mutations E484K et F888L (dans le domaine S2 de la protéine S).
Les premiers cas ont été détectés en décembre 2020 au Royaume-Uni et au Nigeria. Au , 56 cas avaient été découverts au Royaume-Uni. Le Danemark, qui séquençait alors tous ses cas de COVID-19, a découvert 113 occurrences de ce variant du au dont 7 directement liées à des voyages au Nigeria. Au le variant Êta avait été détecté dans 24 pays (pour la France, à Mayotte).
En juillet 2021, il est toujours considéré « variant under investigation », mais il pourrait être converti en « variant of concern » sous réserves d'autres études. Le professeur Ravi Gupta, de l'Université de Cambridge, estime toutefois que ses effets sont dans une certaine mesure prédictibles puisqu'il porte plusieurs mutations déjà observées dans d'autres variants.
Iota (B.1.526)
Le variant B.1.526 dit « new-yorkais » a été observé pour la première fois en sur l’île de Manhattan. En , 3 % des contaminations à New York sont des cas du variant B.1.526. Fin , une augmentation d'au moins 25 % de la présence de ce variant est observée. Des cas à Westchester, dans le Bronx et le Queens, le sud de Manhattan et à Brooklyn sont détectés.
Kappa (B.1.617.1)
Lambda (C.37)
Le variant lambda, également connu sous le nom de C.37, est un variant du SRAS-CoV-2, virus qui cause la Covid-19. Il a été détecté pour la première fois au Pérou en décembre 2020. L'organisation mondiale de la santé (OMS) l'a classé variant d'intérêt le 14 juin 2021. Le génome du variant Lambda présente les mutations d'acides aminés suivantes, toutes présentes dans le code de la protéine Spike du virus : G75V, T761, Δ246-252, L452Q, F490S, D614G et T859N. Il s'agit de mutations faux-sens, c'est-à-dire non silencieuses, induisant un changement fonctionnel. Il est responsable de près de 80 % des contaminations au Pérou.
Mu (B.1.621)
Le variant Mu, également connu sous le nom B.1.621, a été détecté pour la première fois en Colombie en et a été désigné par l'OMS comme variant d'intérêt le . Il a causé des épidémies sporadiques en Amérique du Sud et en Europe.
Anciens variants d'intérêt
ex- Epsilon (B.1.427/B.1.429/CAL.20C)
Le variant CAL.20C ou B.1.429 a été observé pour la première fois par des chercheurs du Cedars-Sinai Medical Center en dans l'un des 1 230 échantillons de virus recueillis dans le comté de Los Angeles. Le variant a été détecté à nouveau dans le sud de la Californie en . En , le variant CAL.20C représentait 36 % des échantillons collectés au Cedars-Sinai Medical Center et, en janvier 2021, le variant CAL.20C représentait 50 % des échantillons.
CAL.20C est le variant présentant une contagiosité plus importante. Il pourrait être à l'origine du nombre exponentiel de cas de Covid-19 : à Los Angeles, plus de 400 000 contaminations supplémentaires ont été enregistrées en un mois du au , alors qu’il avait fallu auparavant dix mois pour atteindre ce seuil. Ce variant comporte trois mutations, dont la mutation L425R située dans le RBD de la protéine spike et qui a été associée à une résistance de certains anticorps neutralisants. Ce variant possède des ressemblances avec celui observé au Danemark (pandémie de Covid-19 au Danemark).
Auparavant considéré comme « variant d'intérêt », Epsilon a été déclassé par l'OMS début .
ex- Zêta (P.2)
L'ex-variant Zêta, ou lignée P.2 (alias de B.1.1.28.2), une sous-lignée de B.1.1.28 — tout comme Gamma (P.1) — a initialement été détecté dans l'état de Rio de Janeiro. Il porte la mutation E484K, mais ni N501Y ni K417T. Il a évolué dans l'état de Rio de Janeiro indépendamment du variant Gamma à Manaus.
Auparavant considéré comme « variant d'intérêt », Zêta a été déclassé par l'OMS début .
ex- Thêta (P.3)
Le variant P.3, dit « philippin », a été découvert dans la région des visayas centrales aux Philippines à la mi-. Il serait présent depuis au moins début . En plus des mutations vues sur les variants B.1.1.248 et P.1, il présente entre autres les mutations E1092K et H1101Y sur la protéine S, dont la signification clinique est incertaine.
Auparavant considéré comme « variant d'intérêt », Thêta a été déclassé par l'OMS début .
Autres variants notables
Variant 20A/EU1 (Espagne)
Le , des scientifiques suisses, américains et espagnols publient l'étude d’un variant du SARS-CoV-2, appelé 20A EU.1. Ce variant semble avoir émergé parmi des travailleurs agricoles en Aragon, dans le nord-est de l’Espagne, « C’est du moins les traces les plus anciennes de sa présence que nous avons pu détecter », précise Emma Hodcroft, l’auteure principale de l’étude et spécialiste de la génétique évolutive humaine à l’université de Bâle, d'après France 24.
Après son développement en Espagne, cette mutation s'est étendue à de nombreux pays européens. Avant le , de très faibles valeurs ont été trouvées en dehors de la péninsule Ibérique. Mais après cela, la mutation s’est propagée en Europe et elle s'est également étendue à la Norvège, à la Lettonie, aux Pays-Bas et à la France. Ce variant s'est même répandu sur d'autres continents, par exemple : la Nouvelle-Zélande ou Hong Kong.
Le variant 20A EU.1 s'est mélangé à la population locale et a ensuite migré vers d'autres régions du pays, représentant alors () plus de 70 % de toutes les séquences analysées par les chercheurs. À partir de la mi-juillet 2020, cette mutation avait rapidement balayé le reste du continent européen, cité plus haut, à commencer par le Royaume-Uni (80 % de toutes les séquences analysées), la Suisse et les Pays-Bas. Emma Hodcroft a souligné qu'en France, « elle représente seulement 40 % des cas analysés, et c’est une autre souche qui est dominante ».
Les mutations génétiques d’un virus sont fréquentes ; elles se produisent lorsque le virus se multiplie dans l'organisme infecté et la plupart du temps elles n’affectent pas son comportement. Mais il n'est pas exclu qu'un nouveau variant s'avère plus virulent.
Le nouveau variant SARS-CoV-2 20A EU.1 s'avère plutôt contagieux, ce qui pourrait expliquer le développement rapide de la deuxième vague. Mais, à ce stade des recherches, rien n’indique qu’il soit plus dangereux que les autres. La chercheuse suisse explique qu'elle n'a jamais étudié un variant de la sorte : « Je n’ai observé aucun variant avec ce type de dynamique depuis que j’ai commencé à étudier les séquences génomiques du coronavirus en Europe ».
Variant 20A/EU2 ou B.1.160
Ce variant a d'abord été dénommé "Marseille 4" par certains auteurs. Initialement détecté en France en et représentant la majorité des séquences en automne 2020, sa fréquence diminue rapidement début 2021 au profit de B.1.1.7.
Variant B.1.1.207 dit « nigérian »
Initialement séquencé en août 2020 au Nigéria — les implications pour la transmission et la virulence ne sont pas claires — mais ce variant a été répertorié comme émergent par les Centres pour le contrôle et la prévention des maladies des États-Unis. Séquencé par le Centre africain d'excellence pour la génomique des maladies infectieuses (ACEGID – African Centre of Excellence for Genomics of Infectious Diseases (en)), ce variant est doté d'une mutation P681H, partagée en commun avec le VOC-202012/01 (variant Alpha) qui a émergé au Royaume-Uni, avec lequel il ne partage aucune autre mutation. À la fin , ce variant représente environ 1 % des génomes viraux séquencés au Nigeria.
Variant B.1.616 dit « breton »
Le variant B.1.616 dit « breton » est détecté en Bretagne fin . Plusieurs malades rattachés à une éclosion au centre hospitalier de Lannion (Côtes d'Armor) présentent les symptômes de la Covid-19, mais avec des tests PCR négatifs. L'Institut Pasteur met en évidence neuf mutations dans la région codant la protéine S : H66D, Del 144/145, D215G, V483A, H655Y, G669S, Q949R et N1187D. La Direction générale de la santé (DGS) le classe comme « variant à suivre ».
La diffusion de ce variant semble limitée. Le bulletin épidémiologique de Santé Publique France du confirme trente-sept cas d'infection par le variant 20C/655Y (trente-quatre en Bretagne et trois dans d'autres régions). La majorité des cas reste liée à l'éclosion du centre hospitalier de Lannion. Les 16 décès dus au variant concernent des patients âgés ou avec des comorbidités. Selon le bulletin épidémiologique, la difficulté à détecter le variant entraine un manque de données concernant sa sévérité et sa transmissibilité.
Variant dit « Henri-Mondor »
L'équipe du laboratoire de virologie de l'hôpital Henri-Mondor AP-HP et l'équipe de la plateforme « génomique » on découvert début 2021 un nouveau variant appelé « variant Henri-Mondor », annonce l’Assistance Publique - Hôpitaux de Paris (AP-HP). Ce variant a été découvert à Créteil, dans une éclosion chez trois professionnels hospitaliers et du conjoint de l'un d'entre eux. « Elle se caractérise par la présence de deux délétions et 18 mutations d'acides aminés, dont 7 à 8 sont localisées dans des positions clés de la protéine « spike », impliquée dans l’entrée du virus dans les cellules ». Dans l'enquête Flash 4 de mars 2021, le variant « Henri-Mondor » représentait 1,8 % des souches séquencées sur le territoire national, on retrouve également les mutations N501Y et L452R que l'on peut observer dans d'autres variants. Les chercheurs ne sont pas en mesure d'affirmer, pour l'instant, si le variant est plus contagieux ou plus létal.
Variant B.1.640 dit « congolais »
Ce variant a été identifié pour la première fois en France en septembre 2021 puis en République du Congo. Il reprend la mutation N501Y des variants Alpha, Bêta et Gamma et s'en distingue par une délétion 137-145Del affectant le domaine N-terminal du virus. Il ne présente pas les mutations d'intérêt E484K, E484Q et L452R qui sont surveillées.
Variant B.1.640.2 dit « IHU »
Ce variant a été détecté dans un hôpital, l’IHU Méditerranée Infection de Marseille le . Il a été identifié sur des patients originaires de Forcalquier (Alpes-de-Haute-Provence). Sa composition génétique amène les scientifiques à penser qu'il s'agirait de l'ancêtre du variant « Congo-Breton » identifié en Bretagne il y a quelques mois.
Pour l'instant, les origines du variant restent encore inconnues, comme son taux de létalité ou sa contagiosité.
Variants et transmission interespèces
Variant B1.1.298 dit « danois »
Début novembre 2020, le variant danois, également appelé ΔFVI-spike par le State Serum Institute (SSI), été découvert dans le Jutland du Nord, au Danemark, et aurait été propagé des visons aux humains via des fermes de visons. Le , il est annoncé que la population de visons au Danemark serait abattue pour empêcher la propagation possible de cette mutation et ainsi réduire le risque de nouvelles mutations. Il a entraîné l'abattage de 15 millions de visons, soit l'intégralité de la population d'élevage du Danemark. Des restrictions de voyage ont été introduites dans sept municipalités du nord du Jutland pour empêcher la propagation de la mutation. Au , quelque 214 cas humains liés au vison avaient été détectés.
L'Organisation mondiale de la santé (OMS) a déclaré que le variant danois a une « sensibilité modérément diminuée aux anticorps neutralisants. » Le State Serum Institute avertit que la mutation pourrait réduire l'effet des vaccins contre la Covid-19 en cours de développement, même s'il était peu probable qu'il les rende inutiles. Il est annoncé le que le foyer de contagion était probablement éteint.
Mutations notables
Les mutations indiquées ci-dessous utilisent la notation : αnnβ.
Cela signifie que le codon en position nn (dans le sens 5' vers 3') a subi une modification de l'un, ou de plusieurs, de ses trois nucléotides. Ainsi, au lieu de coder précédemment l'acide aminé α, il code maintenant l'acide aminéβ. Pour une écriture, une lecture, plus synthétique, les vingt-deux acides aminés sont représentés par une lettre capitale. Ces lettres, ces abréviations, représentent par exemple : l'asparagine par la capitale « N », la tyrosine par la capitale « Y ».
Mutations de la protéine S (spike)
La protéine spiculaire (S) est formée de deux sous-unités : la sous-unité S1, qui contient le domaine de liaison au récepteur cellulaire de l'hôte (en l’occurrence l'ACE2), et la sous-unité S2, qui participe à la fusion de la particule virale avec la membrane cellulaire de l'hôte.
69-70delHV
Cette mutation (également notée ΔH69/V70, ou Δ69-70) est une délétion de deux codons spécifiant des acides aminés : l'histidine en position 69 et la valine en position 70 de la région N-terminale de la sous-unité S1. Elle a été identifiée au Danemark, chez les visons. Dans un isolat cette mutation était associée à trois autres (Y453F, I692V et M1229I), l'ensemble des quatre mutations étant identifié comme ΔFVI-spike.
F140-
La mutation F140-...
Y248*
La mutation Y248*...
L452R
La mutation L452R se trouve dans les variants Delta et Kappa qui ont émergé en Inde puis se sont depuis propagés dans le monde entier. On la retrouve notamment dans les variants Lambda et sous-variants Omicron BA.4 et BA.5. Cette mutation affecte le domaine de liaison de la protéine spiculaire. C'est une mutation significative en ce sens qu'elle améliore la capacité de liaison au récepteur ACE2 et peut permettre d’échapper à certains anticorps.
Y453F
Y453F est une mutation qui affecte un acide aminé impliqué dans l'interaction de la protéine spiculaire avec le récepteur cellulaire du virus, la protéine ACE2. Elle a été identifiée au Danemark, chez les visons. La tyrosine 453 interagit avec l'histidine 34 de la protéine ACE2 ; cette position correspond à une tyrosine chez le vison et les autres mustélidés. Cette mutation est considérée comme une mutation adaptative à l'ACE du vison.
E484K et E484Q
E484K est une « mutation d'échappement » à au moins une forme d'anticorps monoclonal contre le SARS-CoV-2, indiquant qu'il peut y avoir un « changement possible de l'antigénicité ». Les variants B.1.1.248 (Brésil / Japon) et 501.V2 (Afrique du Sud) présentent tous deux cette mutation. Le nom de la mutation, E484K, fait référence à un échange par lequel l'acide glutamique (E) est remplacé par la lysine (K) en position 484.
La mutation E484Q est caractéristique du variant Kappa.
Ces deux mutations seraient des mutations d'échappement aux anticorps (post-infection ou vaccinaux).
N501Y
N501Y indique un changement d'asparagine (N) à tyrosine (Y) en position d'acide aminé 501. Ce dernier est devenu la forme dominante du virus à Columbus fin et et semble avoir évolué indépendamment des autres variants.
D614G
D614G est une mutation qui affecte la protéine de pointe du SARS-CoV-2. La fréquence de cette mutation dans la population virale a augmenté au cours de la pandémie. L'acide aspartique (D) est remplacé par la glycine (G) dans de nombreux variants, en particulier en Europe, mais plus lentement en Chine et dans le reste de l'Asie de l'Est. Il a été proposé que la glycine augmente le taux de transmission, ce qui est cohérent avec des titres viraux et une infectiosité plus élevés vitro. En , il a été signalé que les variants du SARS-CoV-2 dotés de la mutation D614G, plus infectieux, étaient devenus la forme dominante de la pandémie.

Il a été proposé que cette mutation modifie indirectement la conformation du domaine de liaison de la protéine spiculaire, au récepteur cellulaire ACE2. La mutation n'a pas eu lieu dans le domaine de liaison du récepteur, mais elle influencerait celui-ci en rendant sa conformation plus ouverte. Il est affirmé que la glycine, contrairement à l'acide aspartique, ne peut pas faire d’interactions non covalentes avec d'autres acides aminés dans la séquence protéique. Cela aurait pour effet de donner un plus grand intervalle de conformations plus ouvertes et flexibles notamment dans le domaine de liaison du récepteur. La conséquence est que de la liaison de la protéine spiculaire avec le récepteur ACE2 est ainsi facilitée.
D614G est probablement la mutation la plus importante du SARS-CoV-2 pour une autre raison. Les coronavirus peuvent utiliser les récepteurs Fc pour infecter les globules blancs, par un mécanisme connu sous le nom de facilitation dépendante des anticorps. Dans le cas du SARS-CoV-1, il avait été identifié que la séquence activant les anticorps facilitants était l’épitope peptidique « LYQDVNC » localisé sur la protéine S. Cette séquence est présente chez le SARS-CoV-2 et correspond aux codons de la protéine S : L611, Y612, Q613, D614, V615, N616, C617. La mutation D614G ou Q613H empêche l’activation des anticorps facilitants. Le risque est qu’un variant inverse cette mutation et active les anticorps facilitants. Cela pourrait théoriquement être problématique,Interprétation abusive ? sachant que le SARS-CoV-2 est capable de se reproduire dans de nombreux globules blancs comme les macrophages, les monocytes, et les lymphocytes B. Chez le coronavirus du chat, un des rares coronavirus capable de se reproduire activement dans un globule blanc (les macrophages), ce phénomène induit une péritonite infectieuse (PIF). À noter que pour soigner la PIF, le GS-441524, autre nom du remdesivir, s’est montré efficace.
En juillet 2021, quelques rares variants du SARS-CoV-2 circulent avec l’épitope peptidique originel « LYQDVNC ». Il s’agit des lignées A.27 au Burkina Faso , A.28 en Égypte et Jordanie, A.29 en Australie, ou encore la lignée B.6.8 en Papouasie-Nouvelle-Guinée.
Réversion G614D
P681H et P681R
La mutation P681H a été identifiée initialement dans le variant nigérian B.1.1.207. Cette mutation est partagée avec le variant Alpha (dit anglais) et le variant Thêta (dit philippin). Tandis que la mutation P681R est caractéristique des variants Delta et Kappa (dit variants indiens).
Ces mutations interviennent dans la zone de clivage de la protéine spiculaire. La protéine S est constituée de deux sous-unités fonctionnelles : la sous-unité S1 permet la liaison du virus au récepteur de la cellule hôte et la sous-unité S2 assure la fusion de l’enveloppe virale avec la membrane cellulaire. Le codon 681 localisé sur le S1 est à la jonction entre S1 et S2. Le codon 681 se trouve précisément en amont du site de clivage par la furine, localisée entre les codons 682 et 685 de la protéine S (acides aminés RRAR). Il a été proposé que la mutation au codon 681 facilite cette fusion et expliquerait l'augmentation de la charge virale constatée avec le variant Delta. Une hypothèse à vérifier dans la mesure où le variant Kappa (l’autre variant dit indien) a également la mutation P681H mais ne semble pas pour autant aussi contagieux que le Delta.
Le codon 681 est aussi partie intégrante du C-end terminal rule (CendR). Le CendR est un récepteur du SARS-CoV-2 qui peut se lier à la neuropiline 1. Le CendR est libéré après le clivage de S1 et S2. Il est suggéré que la neuropiline 1 facilite le transport axonal du SARS-CoV-2. En d'autres termes, la neuropiline 1 permettrait au SARS-CoV-2 de voyager via les neurones et faciliterait l'infection de nombreux organes. Le CendR intègre le site de la furine et est localisé entre les codons 679 et 685 (acides aminés NSPRRAR). Une mutation au codon 681 pourrait altérer ou améliorer la liaison à la neuropiline 1.
Mutagénèse in vitro
Publiée en janvier 2021, l'étude en prépublication d'une équipe de chercheurs de GSK Vaccines met en évidence in vitro « la capacité incroyable du virus à muter pour échapper aux anticorps » présents dans le plasma d'un patient convalescent, réduisant leur efficacité « à zéro en moins de 80 jours ».
En outre, l'efficacité des vaccins à ARN, induisant « une réponse contre un nombre limité d'épitopes reconnus par les lymphocytes T CD8+ », pourrait être affaiblie par certaines mutations du virus.
Notes
Références
Voir aussi
Bibliographie supplémentaire
- E. Alm, E. K. Broberg, T. Connor, E. B. Hodcroft, A. B. Komissarov, S. Maurer-Stroh, A. Melidou, R. A. Neher, Áine O’Toole, D. Pereyaslov, The WHO European Region sequencing laboratories and GISAID EpiCoV group et N. Beerenwinkel, « Geographical and temporal distribution of SARS-CoV-2 clades in the WHO European Region, January to June 2020 », Euro Surveillance, vol. 25, no 32, (PMID 32794443, PMCID 7427299, DOI 10.2807/1560-7917.ES.2020.25.32.2001410).
Articles connexes
- Virologie
- Maladie à coronavirus 2019 - Pandémie de Covid-19
- Coronavirus lié au syndrome respiratoire aigu sévère (espèce)
- SARS-CoV (coronavirus du SRAS)
- Syndrome respiratoire aigu sévère (SRAS)
- Coronavirus du syndrome respiratoire du Moyen-Orient
- Enzyme de conversion de l'angiotensine 2 (ACE2), porte d'entrée du virus
- Test diagnostique du SARS-CoV-2
- SARS-CoV-2 chez les animaux