Analyse à trois éléments
L'analyse à trois éléments ou analyse à trois taxons est une méthode de reconstruction phylogénétique s'appuyant sur la théorie cladistique. Elle fut introduite par les systématiciens G. J. Nelson et N. I. Platnick en 1991. L'analyse à trois éléments est aussi appelée 3ia (pour "three item analysis") ou TTS (pour "three taxon statements"). Cette méthode s'applique aussi bien aux organismes qu'aux aires biogéographiques dans le cadre cladistique de la biogéographie historique.
Introduction
Dans le cadre de la théorie cladistique, deux méthodes sont aujourd'hui implémentées dans de nombreux logiciels de phylogénie : l'analyse à trois éléments (3ia) et la parcimonie. La méthode 3ia diffère de la méthode de parcimonie par la représentation des caractères, la façon dont procède l'analyse et l'interprétation du résultat obtenu.
La représentation des caractères et hypothèses d'homologie
L'arbre raciné ou hiérarchie
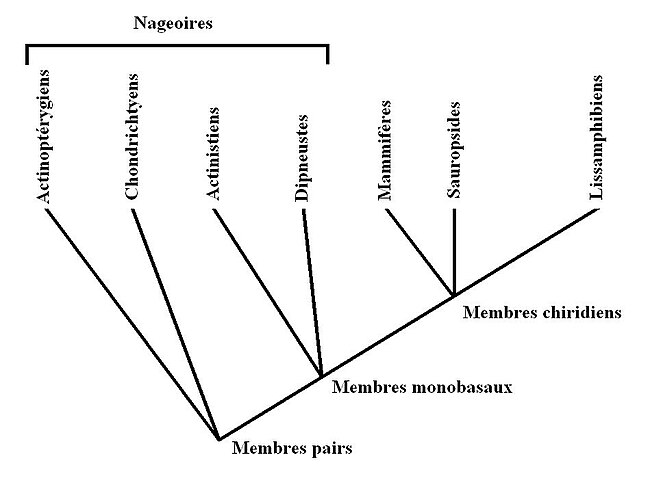
Dans les travaux actuels, les hypothèses d'homologie, que permet de représenter la 3ia, portent sur le phénotype, comme la morphologie, l'anatomie, ou le comportement des organismes (entre autres). Ces hypothèses consistent à hiérarchiser les structures morpho-anatomiques selon leurs états d'existence afin que seuls ceux considérés comme homologues constituent les classes de la hiérarchie. Ainsi, deux taxons auxquels sont attribués le même état homologue appartiennent à la même classe de la hiérarchie et sont eux aussi considérés comme homologues. Si cette hypothèse est retenue sur l'arbre phylogénétique, ils sont considérés plus proches entre eux que de tout autre taxon hors de cette classe. Par exemple, pour une étude sur des vertébrés, les pattes (ou membres chiridiens) peuvent être considérées homologues et ainsi former une classe "membre chiridien". On fait donc l'hypothèse que les taxons présentant de tels membres sont plus proches entre eux qu'ils ne le sont des taxons présentant d'autres membres pairs. Les arbres phylogénétiques consensuels nous apprennent que le cœlacanthe (Actinistien), de par ses nageoires particulières, est plus proche des tétrapodes avec leurs membres pairs que du requin (chondrichtyen). En effet, la caractéristique de l'appendice (membre ou nageoire) des sarcopterygiens (comprenant les tétrapodes, les dipneustes et les Actinistiens, parmi les taxons actuels) est d'être monobasal, c'est-à-dire qu'un seul os le soutient : l'humérus (dans l'appendice antérieur) ou le fémur (dans l'appendice postérieur). Ainsi le concept de "nageoire" n'est plus interprété comme pouvant constituer une classe dans la hiérarchie des hypothèses d'homologie: on dit de ce concept qu'il est paraphylétique.
Dans le schéma précédent, l'inclusion de l'état "membres monobasaux" dans la classe dite "membres pairs" indique que cet état est supposé informatif et donc pertinent pour le regroupement des taxons. Cela revient à dire que l'état "membres monobasaux" est considéré comme apomorphe parmi les "membres pairs". Cette proposition doit pouvoir être justifiée par un(des) critère(s), y compris le critère extra-groupe, utilisant un ou des taxons du groupe externe. Ainsi, l'utilisation d'un taxon du groupe externe lors de l'analyse (en vue d'un enracinement a posteriori) est inutile puisque l'enracinement des hypothèses est réalisée en amont de l'analyse. Le test des hypothèses est effectué par l'analyse de reconstruction de l'arbre phylogénétique. Il aboutit au rejet ou non des états proposés comme pertinents. Si un état est rejeté, il est interprété être une homoplasie. Au contraire, s'il est retenu sur l'arbre phylogénétique, il est interprété être une synapomorphie. Les états supposés pertinents et retenus en tant que synapomorphies permettent la caractérisation des taxons. On remarque alors l'équivalence entre taxons et états de caractères (cf schéma): ici, la classe "membres pairs" correspond au taxon Gnathostomes, "membres monobasaux" au taxon sarcoptérygiens et "membres chiridiens" au taxon tétrapodes.
Données manquantes et données non applicables
L'analyse à trois éléments (3ia) n'utilise pas la représentation des caractères sous forme de matrice taxons-caractères comme la méthode de parcimonie; la 3ia représente les caractères sous forme d'arbres enracinés. Alors qu'il est nécessaire de remplir toutes les cases d'une matrice, y compris pour les taxons non renseignés (on doit alors insérer un signe approprié, comme un point d'interrogation), l'arbre raciné permet au contraire de ne pas prendre en compte de tels taxons dans les caractères. En réalité, un taxon non renseigné pour un caractère peut l'être pour deux raisons : soit à cause d'un doute sur l'état qu'il faudrait lui attribuer, soit de par la non-pertinence de chacun des états (dans le cas où le taxon ne présente pas la structure morpho-anatomique dont il est question par exemple). La prise en compte de ces deux cas en 3ia est différente, permettant une claire distinction entre le premier pour lequel tous les états restent possibles, et le deuxième pour lequel aucun des états n'est souhaitable.
L'arbre de droite est un exemple de la représentation d'une relation homologie pour 6 taxons:
taxon A: présentant l'état "antennes absentes".
taxons B et C: présentant l'état "antennes recourbées".
taxons D et F: présentant l'état "antennes en forme de massue".
taxon E: tête non préservée, donc manquante
Pour le taxon A, aucun des états "antennes en forme de massue" ou "antennes recourbées" n'est applicable puisque ce taxon ne possède pas d'antennes. Pour le taxon E, on peut imaginer un spécimen de collection dont la tête aurait été égarée ou bien un fossile pour lequel la tête n'aurait pas été préservée. Lors du replacement de ces taxons dans une des classes de la hiérarchie correspondant à une hypothèse d'homologie, le taxon A ne peut pas être inclus dans une classe "présence d'antennes". Il faut d'ailleurs reporter l'information selon laquelle il ne peut appartenir à aucun des états proposés : pour ce faire, il est attribué à une classe plus générale que toutes les autres c'est-à-dire non informative pour le regroupement des taxons lui étant assignés. Par contre, pour le taxon E, l'information sur son état n'est pas en notre possession. Il convient alors de n'assigner ce taxon à aucune des classes. Il reste absent du caractère.
L'analyse
Lorsqu'une analyse phylogénétique est réalisée selon la méthode 3ia, tous les caractères sont décomposés en assertions minimales à trois taxons ("3is" en anglais, pour three-item statement). Une telle assertion indique que deux taxons sont plus proches entre eux que d'un troisième. Étant donné qu'il est toujours possible de dire de deux taxons qu'ils sont apparentés, un 3is ne peut pas porter sur moins de trois taxons, d'où son nom.
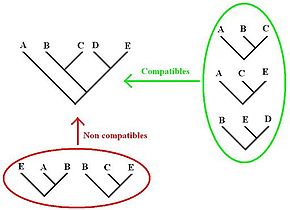
La reconstruction de l'arbre phylogénétique recherche le plus grand ensemble de 3is compatibles. Ces 3is sont alors combinés pour reconstruire l'arbre optimal, le critère d'optimalité étant ici la congruence maximale.
Soient les taxons A, B, C, et D. Soient les 3is suivants : (A(B,C)) (B(C,D)) (A(D,C)) (C(B,D)). Les 3is (B(C,D)) et (C(B,D)) sont incompatibles entre eux puisqu'ils n'indiquent pas la même relation entre les trois mêmes taxons B, C et D. Cependant, ils restent compatibles avec d'autres 3is de la liste.
- 1er ensemble de 3is compatibles : (A(B,C)) (B(C,D)) et (A(D,C)). Le premier et le dernier nous indiquent que les taxons B,C et D sont plus proches entre eux que de A. Il est possible de les combiner afin de reporter cette information en une seule inclusion : (A (B,C,D)). Cependant, le deuxième 3is de la liste nous indique que parmi B, C et D, les taxons C et D sont plus proches entre eux que de B. Il suffit de reporter cette information sur l'inclusion précédente : (A (B (C,D))).
-2e ensemble de 3is compatibles : (A(B,C)) (A(D,C)) et (C(B,D)). Les deux premiers indiquent que les taxons B, C et D sont plus proches entre eux que de A. Leur combinaison aboutit ainsi à (A (B,C,D)). Le troisième 3is de la liste précise que parmi B, C et D, les taxons B et D sont plus proches entre eux que de C. Cette information prise en compte, on obtient : (A (C (B,D)).
Cet exemple aboutit à deux ensembles des 3is aussi grand l'un que l'autre. Les deux arbres obtenus à la suite de la combinaison de ces deux ensembles de 3is sont donc équi-optimaux.
La 3ia recherche alors quelles sont, parmi les arbres équi-optimaux, les relations jamais contredites. Ici, il s'agit de (A(C,B,D))qui est alors proposé comme le résultat de l'analyse phylogénétique.
Plusieurs programmes informatiques ont été développés afin de combiner des listes de 3is. Cependant, le seul permettant une reconstruction phylogénétique à partir des 3is obtenus par la décomposition de caractères représentés sous la forme d'arbres enracinés est le logiciel LisBeth développé par une équipe du Muséum national d'Histoire naturelle de Paris. Il est aussi le seul logiciel de 3ia toujours en développement.
Voir aussi
Autres références
Bibliographie
- I. J. Kitching, P. L. Forey, C. J. Humphries et D. M. Williams, "Cladistics. Second edition. The theory and practice of parsimony analysis", Oxford science publications, 1998.
- G. J. Nelson et N. I. Platnick, "Three-taxon statements: a more precise use of parsimony?" Cladistics, 7, 351-66